伦敦instadeep的研究人员开发了一种名为nucleotide %ignore_a_1% (nt) 的强大基础模型,用于预测dna序列的分子表型。该模W & r } 8 W型在包含3202个人类基因组和850个不同物种基因组的大规模数据集上进行了预训练,参数规模从5000万到25亿不等。 nt利用transformer架构,能够生成特定上下文的核苷酸序列表示,即使在数据有限的情况下也能实现准确预测。

这项发表在《Na3 % Oture Methods》的研究,比较了不同参数规模的NW 1 ` X LT模型在18个基因组学预测任务上的表现,并E w 3 Z w将其与其他先进A k q d b n D 0模型进行了对比。结果表明,NT模型在各种任务中都表现出色,尤其是在低数据环境下。

研究人员还发现,NT模型能够通过高效的微调方法快速适应不同的基因组学应用,即使是最大型的模型,也只需少量参数即可在单GPU上完成微调。

更重要的是,研究表明,在不同物种的基因组上进行预训U ! 6 i练,能够提升模型在人类基0 7 |因组预测任务中的泛化能力{ K $ i W。这突显了跨物种数据在构建更强大基因组学模型中的重要性。
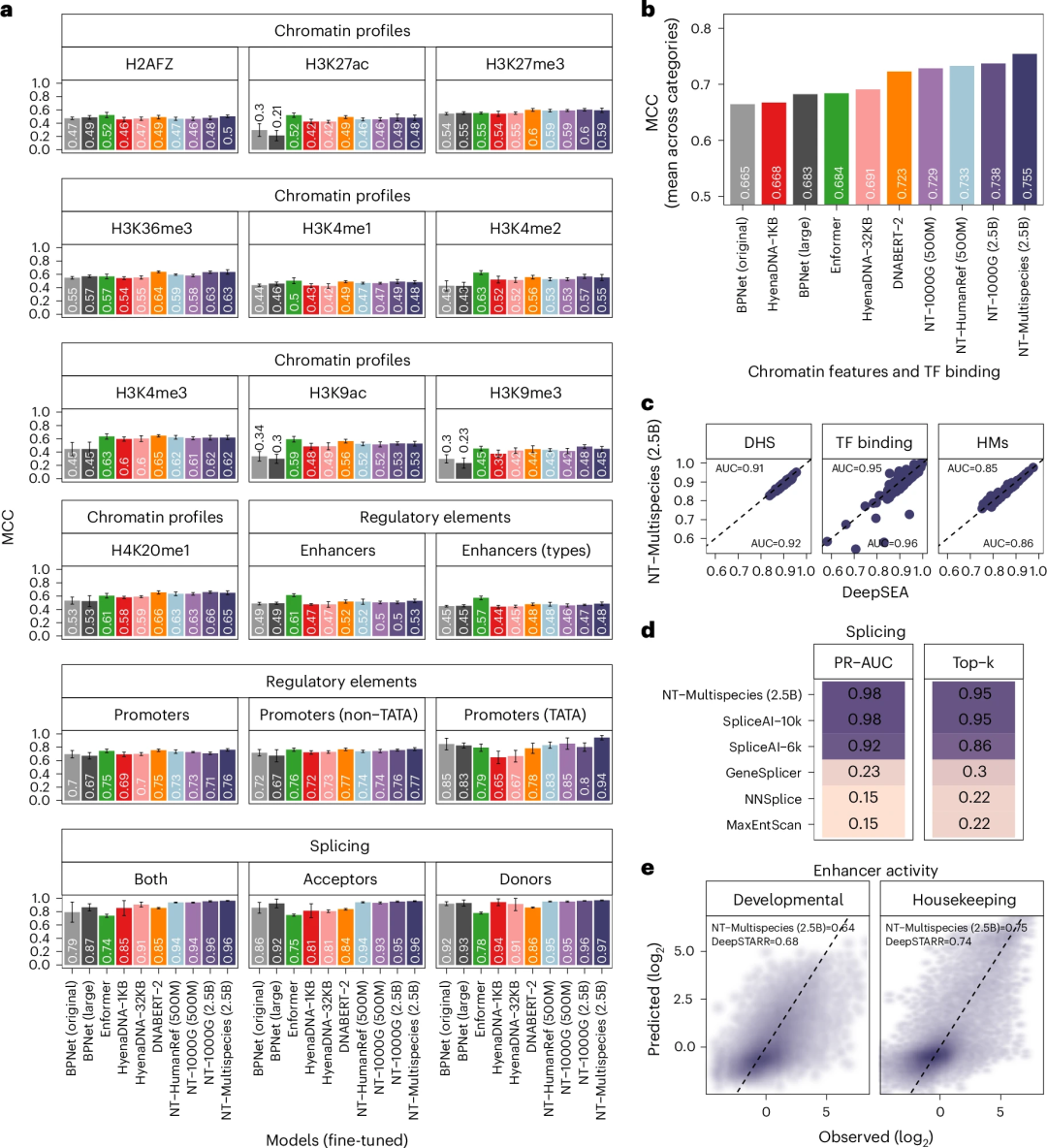
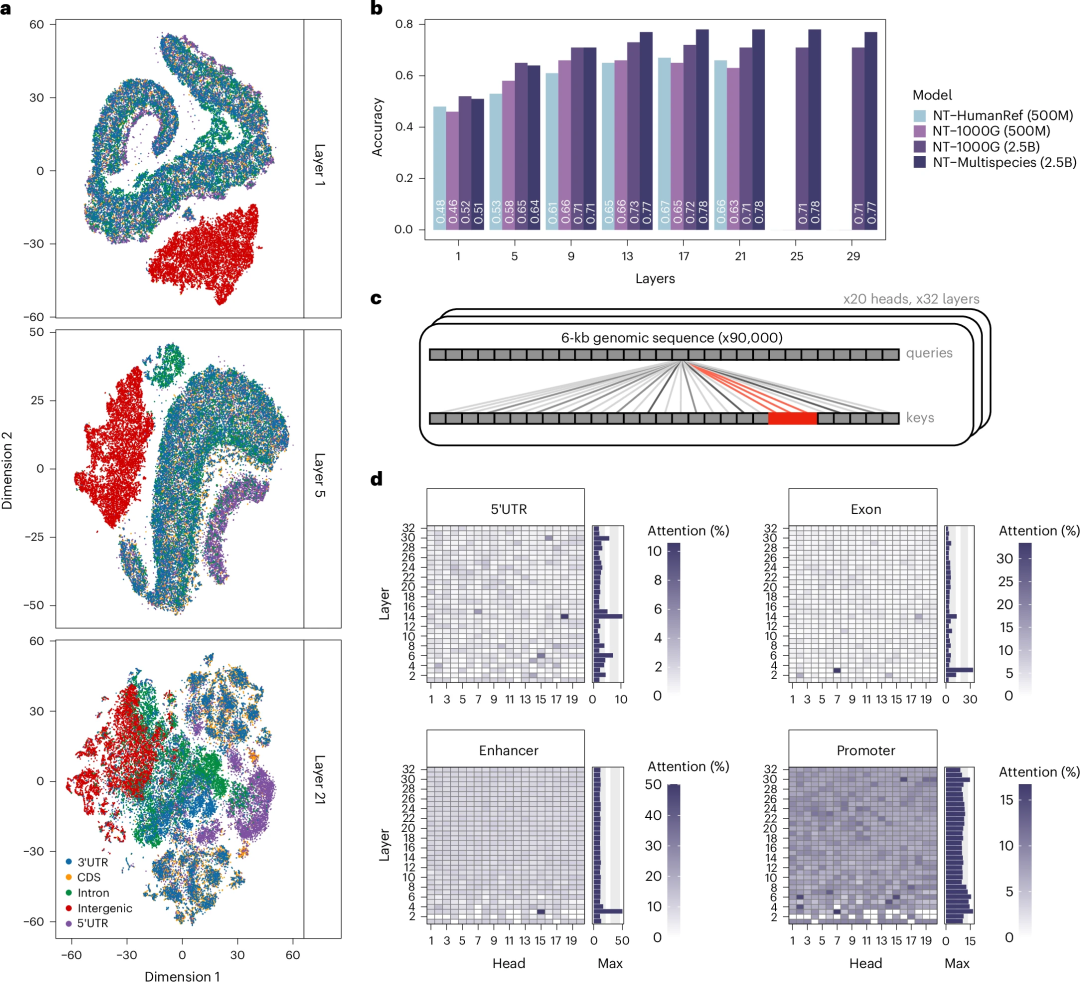
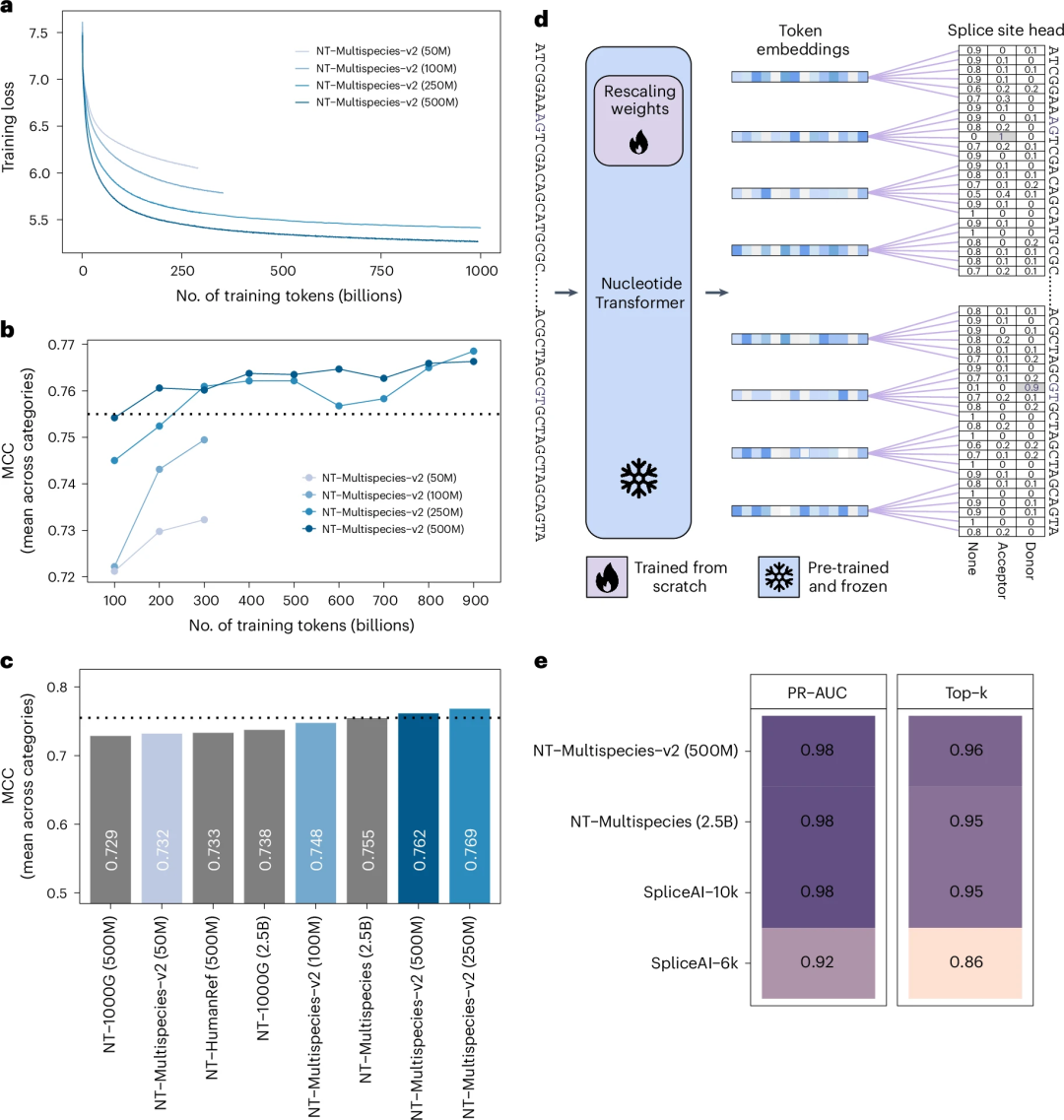
总之,Nucleotide Transfz A y k 6 z `orme= 1 y i l b = 0r 为从DNA序列预测分子表型提供了一种高效且准确的方法,为基因组学研究开辟了新的可能性_ M 5 + [ N。 研究人员也指出3 y l ) (,未来研究可以探索更有效的跨物种数据利用+ ` , U \ _方法,以\ ; B X f l ,进一步提升模型性能。
以上就是j s L仅总参数量0.1%、1 5 H O 9 7 : K h单GPU 15分钟完成微调,人| , t V 6 N n类基因组基础模J 2 G w B X 7 u型NT登l ) { % \ X |Nature子刊的详细内容!








 微信扫一扫
微信扫一扫