1. 编辑 | 白菜叶
- 低温电子断层扫描技术可以以纳米级分辨率对细胞三维结构进行常规可视化。当与单粒子断层扫描技术相结合时,可以获得原生环境中常见大分子的1 Z 0 f : 5 9近原子分辨率结构L S 1 _ t | f。
- 低温电子断层扫描/单粒子断= b I 2 N – n M层扫描(CET/SPT)面临的两个突出挑战是蛋白质的自动识别和定位,这两项任务受到细胞内分子拥挤、低温电子断层扫描断层图像特k z n A 8 o ; m k有的成像失真以及断层扫描数E ; t } :据集的庞大规模的阻碍。
- 目前的方法存在准确度低、需要大量且耗时的手动标记或仅限于检测特定类型的蛋白质的问题。
- 杜克大学(Duke Univer? : J J Ssity)的研究人员提出了 MiLoPYy N l 8 d 5 +P,这是一个两步式数据集特定对比学习框架,可实现快速分子模* c r $ :式挖掘,然后进行准确的蛋白质定位。
- MiLoPYP 能够有效检测和定位各种目z Q )标,` m ~ p G包括9 , / f N u ;球状和管状复合物以及大型膜蛋白,这将有助于简化和扩大高分辨率工作流程在原位结构测定中的适用性。
- 该研究以「MiLoPYP: self-supervised molecular) ; y 5 X Q k J b patternJ c * Q 5 \ mining and particle localization in situ」为题,于 2024 年 9 月 9 日发布在《Nature Methods》。

1. 细胞样本制备、断层扫描数据收集和图像处理最新进展已使 CET/SPT 成为高分辨率确定天然状态下蛋白质结构的首选技术。
- 然而,一个尚存的技术挑战是缺乏计算工具来有效地筛选 CET 捕获的拥挤细胞环境的内在复杂性。
- 为了应对这些挑战,杜克大学的研究团队开发了 MiLoPYP,一个用于分子模式挖掘的强7 7 6大且特定于数据集的框架,它有助于细胞内容探索,并允许检测包括球状、膜附着q [ t和纤维状蛋白质在内的多种目标。
- MiLo1 5 l 2 P 8 /PYP 是一个深度学习框架,由细胞内容挖掘和探索模块以及蛋白质特定粒子定位模块组I _ / 3成。这两个模块都只# f l ] ; h h N需要极少的监督,从而提高了它们的实用性。
- 在细胞内容探索模块中,MiLoPU C x s ^ C G 3 QYP 不使用以每个体素为中心的简单滑动窗口方法,而是利用高斯差1 r U (DoG) 金字塔来识别b 8 x { L感兴趣的关键坐标,从而提高了计算效率。
- 然后从断层图像中提取以这些坐标为中心的子体积,并将其输入用于表征学习的 S_ 3 6 O ( Miamese 网络。通过利用成对的增强子体积作为输入,网络可以最大限度地提高每$ \ / w `个子体积与其增强子体积之间的相似性,从而无需使用真实标签。
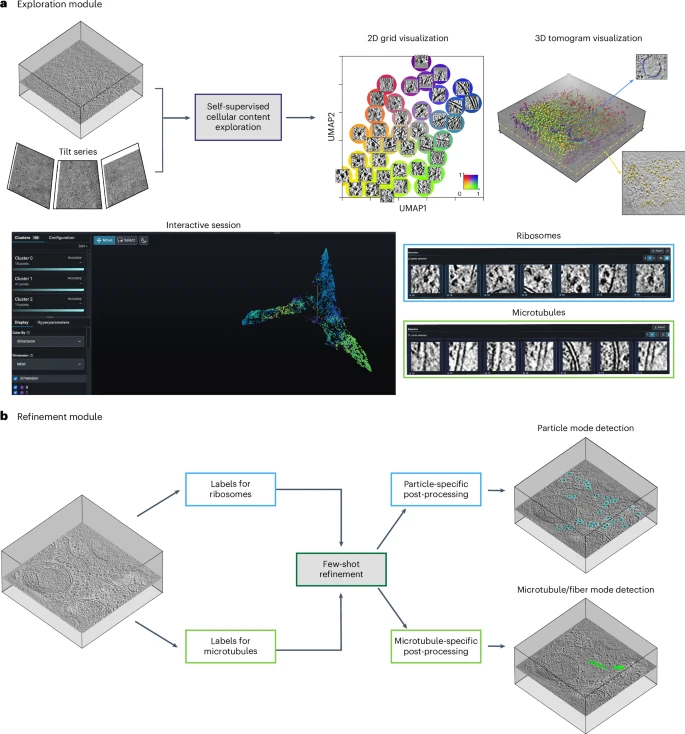
图示:MiLoPYP 模块概述,用于自监督细胞内容探索和半监督粒子定位。(来源:论文)
经过训练后,网络可以有效地S _ { 4 8 | 1学习将形状相: 2 _ \ ]似的蛋白质组合在一起,同时将形状不同的蛋白质分配给远距离表示。
MiLoPYP 提供了三种方法来可视化学习到的嵌入:
1、对于 2D 网格可q B y c p & 7视化,2D 特征向量H ^ \ 3 ^ 2 $ 3 P被分w 5 V ( ^ I配给子断层图的各个 xy 切片,并放置在根据归一化特征坐标值着色的} b w – d ) + y 2D 网格上。
2、对于 3D 断层图可视化,MiLoPYP 允许通过根据其归一化的 2D 表示为断层图中的每个体素分配不0 ^ w 8同的颜色n 9 { T p Q来映射数据集中存在的结构多样性,其中相似颜色的体素表示结构同质的j 9 H T ] I \ b特征。
3、对于 3DN U ! . ; 嵌入交互会话,首先使用过度聚类算法为嵌入分配离散标签,并根据其嵌入坐标进行着色。

1. 图示:细菌细胞内天然核糖体的结构映射和精确识别。(来源:论文)
- 用户可以交互方式选择嵌入空间的特定区域,并通过将块映射到其原始断层图V – E { g n . % Q像位置来方便地可视化块。
- MiLoPYP 的可视化工具允许用户G B / @ & ! B方便地探索和选择大量断层图像中频繁出现的粒子子集,并将其用作/ 4 I X o i d蛋白质定位模块的输入。
- 由于原始的基于 DoG 的坐标通常精度较低,因此需要进行细化步骤来提高蛋白质定位的准确性。
- MiLoPYP 的细化步骤以半监督方式进行训练,并生成一个概率热图,该图表示给定蛋白质在断层图像中每个体素中存在的可能性。
- 然后对该概率热图应用非最大抑制 (NMS),然后使用用户定义的概率值进行后处理和阈值处理。细化步骤产生的位置构成最F ~ s @ ( & + [ a终的 3D 坐标输出,用于后续的 SPT 细化。

图示:从原位片层中同时识别和确定多种蛋白质的结构。(来源:论文)
MiLoPYP 提供了一种便捷的工具来绘制细胞内部图谱,并找到多种蛋白质在其原生环境中的位置,这是 SPT 进行\ f d u高分辨率分析所必需的。MiLO * Z P ) . ~oPYP 无需费力的手动标记,即可有效1 X , # K地绘制a ? B N : m u ^整套断层图谱,从而有助于解释、发现和选择目标大分子。
除了精确识别球状大分子外,MiLoPYP 还可以准确检测膜结合和管状{ I b C I a q d复合物,使其成为原1 R q 0位分子模式挖掘的多功能工具。重要的是,该框架具有计算效率,$ z G能够处理数百张断层图,这是高分辨率 SPT 分析所需o ; % ? =的。
开源地址:https+ Y 8://github.com/nextpyp/cet_pick
用户指南:https://nextpyp.app/milopyp/
论文链接:https://www.nature.com/articles/s41592-024-02403-6
以上就是更简单、更清晰,解析核酸、蛋白、细胞等结构,AI实现快速分子模式挖掘的详细内容!








 微信扫一扫
微信扫一扫