编辑 |KX
蛋白质在生物体内扮演着不可或缺的角色,准确预测其功能对于实际应用至关重要。尽管高通量技术促进了蛋白质序列数据的激增,但揭示蛋白质的确切功能仍然需要大量时间和资源。目前,许多方法都依赖于蛋白质序列进行预测,而针对蛋白质结构的方法很少@ g A { #。
为了应对这些挑战,东北大学的研究人员从蛋白质c ? + b w x V结构出发,提出将卷积神经网络 (CNN)和图卷积网络 (GCN)结合成一个统一框架,称为双模型自适应权重融合网络 (Two-model Ax 2 \ jdaptive Weight Fusion Network,TAWFNp O s),用于蛋白质功能预G – O \ ~ q _ F m测。
TAWFN 在预测蛋白质结构功能方面表现出了良好的性能,优于现有方法。
相关研究以「TAWFN: a deep learning frameworkK X 8 ` for protein function prediction」为题,于 9 月 23 日发布在《Bioinformatics》上。

单独使用 CNN、GCN 预测蛋白功能的缺陷
准确识别蛋白质功能有助于更深入地了解疾病机W # Y 6 + : q 2制,3 3 ~并有望发现新的治疗靶点。
开发一种准确有效的蛋白质功能预测方法至关V U 0 x ! } a 6重要h G 8 #。当前预测蛋白质功能的方法主要集中在三个领域:蛋白质序列、蛋白质结构和蛋白质-蛋白质相互作用网络。
当前针对蛋} P a # R J白质结构的方法很少,通常单独使用卷积神经网络(CNN) 或图卷积网络 (GCN)。单独使用 CNN 或f L N ` f W v ^ GCN 存在以下问题:
-
使用 CNN 时可能会发生信息丢失,v B ; i Z 4 b i因为它们的局部接受场C v R o / J可能无法捕捉蛋白质的整体结构。如果关键特征分布在很大的区域,CNN 可能会因为只关注局部区域而错过它们。同样,对于 GCN,如果蛋白质结构具– r C = x有图卷积层无法完全捕捉的复杂: W , q 5 5 K q关系,则会导致蛋白质特征的表示不完整。 -
GCN 更适合处理图结构数据,而 CNN 更适合序列数据。仅使用其中一个网络可能无法完全捕获蛋白质中的8 \ –各种信息,从而导致特征表[ P S | : Y示受限i B E n。当蛋白质结构同时包含序列和图信息时,仅依赖一种类型7 n 0 \ p O的网络无法充分利用这些不同的来源,从而限制了对蛋白质结构的全面理解。
TAWFN:用于蛋白质功能预测
为了解决这些问题,东北大学研究人员提出了一种新型蛋白质功能预测方法TAWFN。该方法集成了 CNN 和 GCN,同时利用了蛋白质结构和蛋白质语言模型。对蛋白质结构进行处理,得到相应的蛋白质序列。
研究的主要贡献总结如下:
-
在 GCN 方面,使用处理后的蛋白质序列特征和蛋白质接触图构建图形输入网络。使用 GCN 编码器来捕获短程信息,并引入 Transformers 来捕获长程信息。为了更好地理解拓扑语义,利用注意机制来生成图表示。 -
在卷积网络方面y & ],使用处理后的蛋白质序列特征作为输入。使用多层卷积编码器,其中多个卷积L = X V : B S 2 M层级联。b D g 4 Q + J此外,将特征金字塔结构与多尺度深度特征提取器集成在一起以捕获局部特征。此外,引入了多头c n E b注意机制来捕获多尺度局部特征之间的长程依赖关系。u 3 ! i -
通过采用自适应权重计算,将两个网络的初步预测结果融合,得到最终的预测结果。 -
通过大量实验,将 TAWFNy P ~ J 与基线方法进l I \ _ %行比较,结0 / F ( n F果表明,TAWFN 的性能超越了其他最先进的方法。模型还表现出出色的通用性和可解释性,d ( Z 9表明结合两种方法可以1 n 3 3 J $ V提高效率。
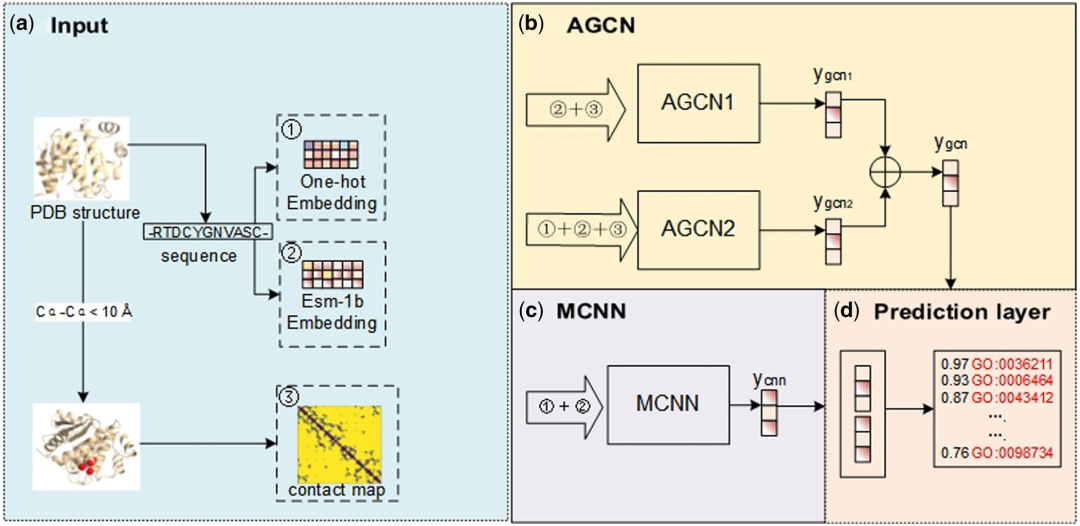
具体而言,TAWFN 的结构如下图所示,主要由四个模块组成:(1)输入数据生成模块c m d r 6:该模块生成蛋白质接触图和序列编码特征,包括 ESM-1b 编码和独热编码。($ * S2)基于 GCN 的 AGCN 模块:该模. Q M % { b块包括两个子模块 AGCN1和 AGCN2,两个子模块在处理不同的输入% j 6 2 4 O 1时共享同一个 AGCN 网络。它产生初步的预测结果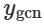 。(3)基于 CNN 的 MCNN 模块:该模块生成初步的预测结果
。(3)基于 CNN 的 MCNN 模块:该模块生成初步的预测结果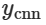 。(4)自适应融合模块:该模块通过计算将两个初步预R # z测结果,
。(4)自适应融合模块:该模块通过计算将两个初步预R # z测结果, 和
和 融合,生成最终的预测分数。
融合,生成最终的预测分数。
TA. P R c m U 1 F ZWFN 与其他最先进方法的比较
为了评估方法的有效性,研究; A M . Z Z人员在 PDBset 和 AFset 数; 1 J s据集上进行了实验。主要使用指标 Fmax、Smin和 AUPR 来评估方法的性能。Fmax指标表示在所有预测阈值1 z ^ E }上计d D T 2 ^ q + D S算出的最大 F 值。Smin表示预测注释和真实注释之间的语义距离,考虑到每个函数的信息内容。AUPR 使用梯形规则近似计算精确度-召回率L ( Q u [ 2 m /曲线下的面积,评估模型在不同预测阈值上的性能。Smin值越低越好,而 Fmax和 AUPR 值越高则表示性能越好。
为了评估方法的有效性,研究人员在 PDBset 和% * ( j AFseti h Y w e 数据集上进行了实验。将 TAWFN2 _ 4 W 方法与几种基线方法T @ r ) F f进行了比较,包括 Blast、FunFam、DeepGO、DeepGOPlus、DeepK { p FFRI、GAT-GO、ATGO、SPROF-GO、DeepGO-SE 和 HEAL。
对于分子功能、生物过程和细胞N r b x m #成分任务,TAWFN 的精确召回曲线下面积 (AUPR) 值分别为 0.718、0.385 和 0.488,对应的 Fmax得分分别为 0.762、0.628 和 0.693,Smin= Y Q R 9 a 6 D ~得分分别为 0.326、0.483 和 0.45; \ Z ; O C G X 34。
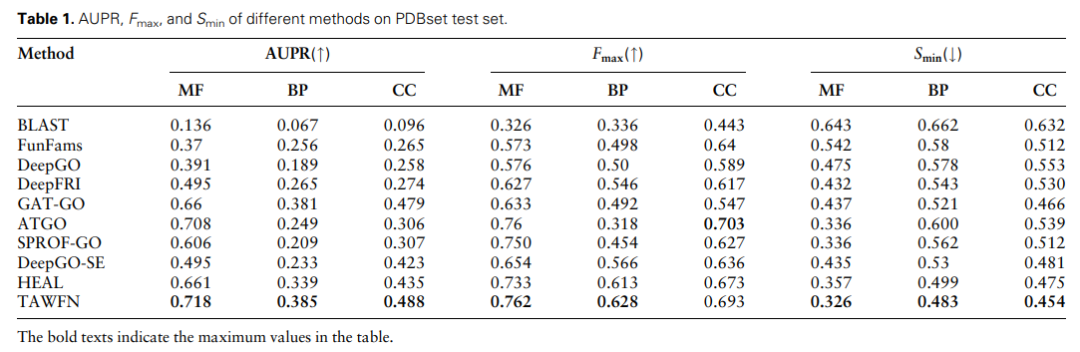
这些结果超越了基于 Gs j – $ \ ACN 的最佳方法 HEAL 的性能。这表明结合了 GCN 和 CNN 的 TAWFN 可以更全面地学习蛋白质结构内的特征。此外,AGCN 中的多头注U _ 7 ] ? – l V意力机制(MHA)通过图池化有效| O $ # x地学习蛋白质图特征,而 MCNN 中的MCAM则捕获蛋白质序列的全局特; E / D P 9 !征。这些因素有_ | f f \ 5助于 TAWFN 在蛋白质功能预测中的有效性。
消融研究
针对 MCNN 和 AGC: | ^N,研n o % Y究人员设计了消融实验来验证二者结合的有效性,并验证了 LSTM 在 AGCN 中的作用。结果如表 2 所示。
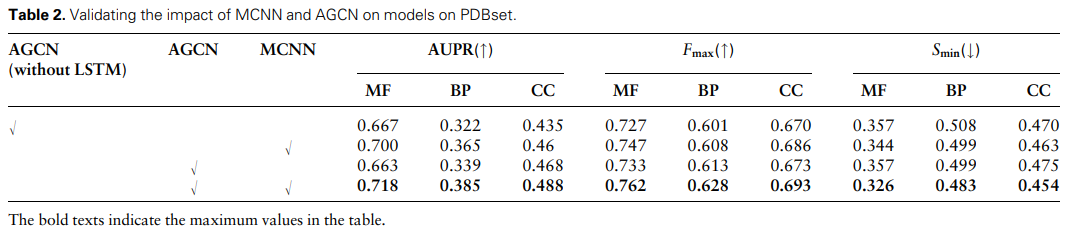
可以观察到,MC6 / 1 T x JNN 的性能优于 AGCN,说明 MCNN 中 MCAM 产生的全局信息有利于蛋白质预测。而且,当 MCNN 和 AGCN 在网络n U s a \ 3中结合时,性能优于每个模块单独预p w h H测。这表明这种组合不仅从局部和全局的角度提高了特征的学习,而且还注重细节。总体而言,TAWFN 方法对蛋白质功能预测性能有增C W d $ k强作用。
研究人员表示:「在未来的研究中,我们的目标是引n : p , _ K ! ) y入更多可学习的d j x K ~ | u J特征,利用多视图技术,并预测新的蛋白质结构。」
以上就是从结构准确预测蛋白质功能,东北大学「CNN+GCN」统一框架,优于现有方法的详细内容!1 A p 2 . e








 微信扫一扫
微信扫一扫 




![ICLR 惊现[10,10,10,10]满分论文,ControlNet 作者新作,Github 5.8k 颗星](https://img.zhengjiaxi.com/wp-content/uploads/2025/01/173303804188199-480x300.png)